Learn
About Us
The university that empowers ingenuity, innovation, and entrepreneurship.
Our Academic
Offer
With our active learning methodology, our students experience engineering from day one.
Ingenuity,
dedication, and
the pursuit
of excellence
Our students' DNA: achieving highest academic achievement and personal development.
International
alliances
We've built strong relationships with the best educational institutions in the world.
Research
Experience
The right path to finding better solutions.
Sustainability UTEC
Sustainability documents
Contact:
Giancarlo Marcone
HACS DIRECTOR
gmarcone@utec.edu.pe
Learn
About Us
The university that empowers ingenuity, innovation, and entrepreneurship.
Our Academic
Offer
With our active learning methodology, our students experience engineering from day one.
Ingenuity,
dedication, and
the pursuit
of excellence
Our students' DNA: achieving highest academic achievement and personal development.
International
alliances
We've built strong relationships with the best educational institutions in the world.
Research
Experience
The right path to finding better solutions.
Sustainability UTEC
Sustainability documents
Contact:
Giancarlo Marcone
HACS DIRECTOR
gmarcone@utec.edu.pe
You are here
Simulación Dinámica Molecular Usando microscopio computacional para lucha contra el COVID-19
Autor:

Prof. Paul Cardenas.
Departamento de Bioingeniería
pcardenas@utec.edu.pe
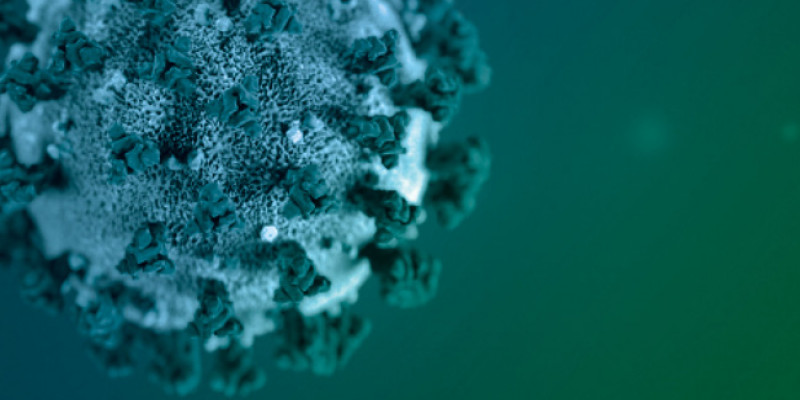
El síndrome respiratorio agudo severo coronavirus 2 (SARS-CoV-2) es el virus detrás del brote mundial de la pandemia del COVID-19. El SARS-CoV-2 es un virus de estructura esférica que mide aproximadamente 100 nanómetros (nm) (Figura 1). Una de las características importantes del SARS-CoV-2 es la presencia de proteínas spike (S) que permiten que estos virus ingresen en las células huésped y provoquen una infección. El coronavirus SARS-CoV-2 ingresa a la célula mediante la interacción con la proteína S que es una proteína que se encuentra altamente glicosilada y que contiene alrededor de 1400 aminoácidos. La proteína S contiene dos subunidades (S1 y S2) funcionales que se dividen en tres dominios: cabeza, tallo, y cola citoplasmática. La subunidad S1 N-terminal forma la cabeza globular de la proteína S y la subunidad S2 C-terminal--que es el elemento más conservado de la proteína S--forma el tallo de la proteína y está directamente incrustada en la envoltura viral (Figura 2). La subunidad S1 reconoce y se une a los receptores en la célula huésped, mientras que la subunidad S2 es responsable de fusionar la envoltura del virus con la membrana de la célula huésped. La subunidad S1 contiene el motif de unión al receptor (RBM) que es responsable de la interacción con la enzima convertidora de angiotensina 2 (ACE2). Esta interacción entre RBM y ACE2 es el punto de ingreso en la patogénesis de la infección.
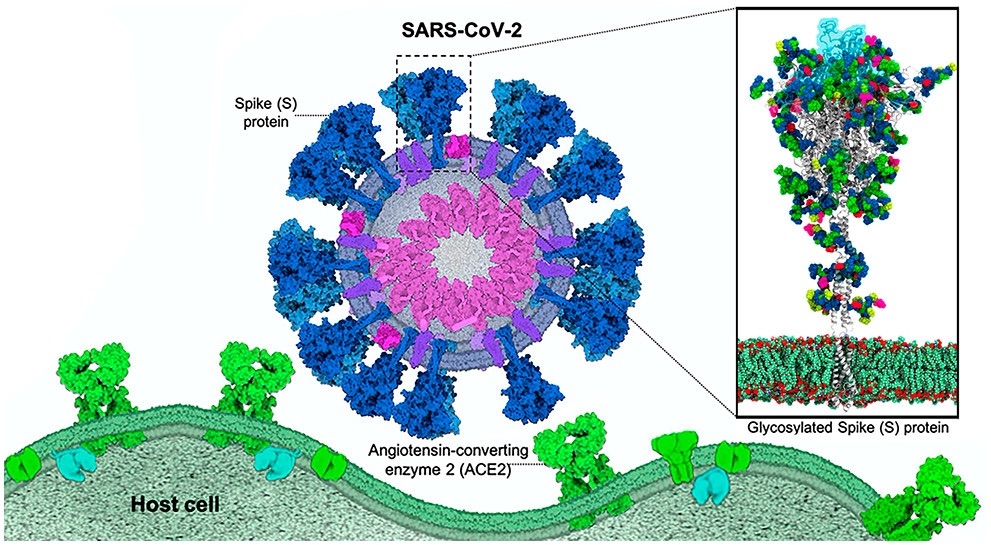
Figura 1. El SARS-CoV-2 es el virus detrás del pandemia mundial global. El SARS-CoV-2 es un virus de estructura esférica que mide aproximadamente 100 nanómetros (nm) y contiene la proteína spike que tiene el motif de unión al receptor (RBM), el cual es responsable de la interacción con la enzima convertidora de angiotensina 2 (ACE2). La interacción entre RBM y ACE2 es el punto de ingreso del virus [2].
Fuente. Pablo R,Arantes; Aakash Saha; Giulia Palermo. ACS Publicatons. (2020)
Cuando la subunidad S1 se une a los receptores de la célula huésped, deben ocurrir dos cambios conformacionales dentro de la subunidad S2 para que se complete la fusión del virus con la membrana celular. La primera conformación (pre-hairpin) implica la transformación de un linker no estructurado en la subunidad S2 para convertirse en helicoidal. El segundo cambio conformacional implica la inversión de las C-hélices de esta subunidad en un coil, lo que da como resultado la formación de un haz de 6 hélices. Una vez que ocurren los cambios conformacionales, el péptido de fusión se ancla a la membrana de la célula permitiendo que el virus se acerque a la membrana celular y finalmente entregue la nucleocápside viral a la célula. Sin la proteína S, el nuevo SARS-CoV-2 no puede interactuar con las células de huéspedes de animales y humanos, por lo tanto, la proteína S representa un objetivo ideal para los esfuerzos de investigación de vacunas y antivirales. Estudios experimentales han demostrado que la mutación D614G en la subunidad S1 aumenta la infectividad. Se entiende molecularmente que S1 es el núcleo en la ruta de infección y, por lo tanto, sus superficies pueden ser usadas potencialmente para el desarrollo de vacunas. Las proteínas S están recubiertas con una capa de azúcar que presenta 22 N-glucanos por monómero que le ayuda al camuflaje para evadir la respuesta inmune de la célula huésped.
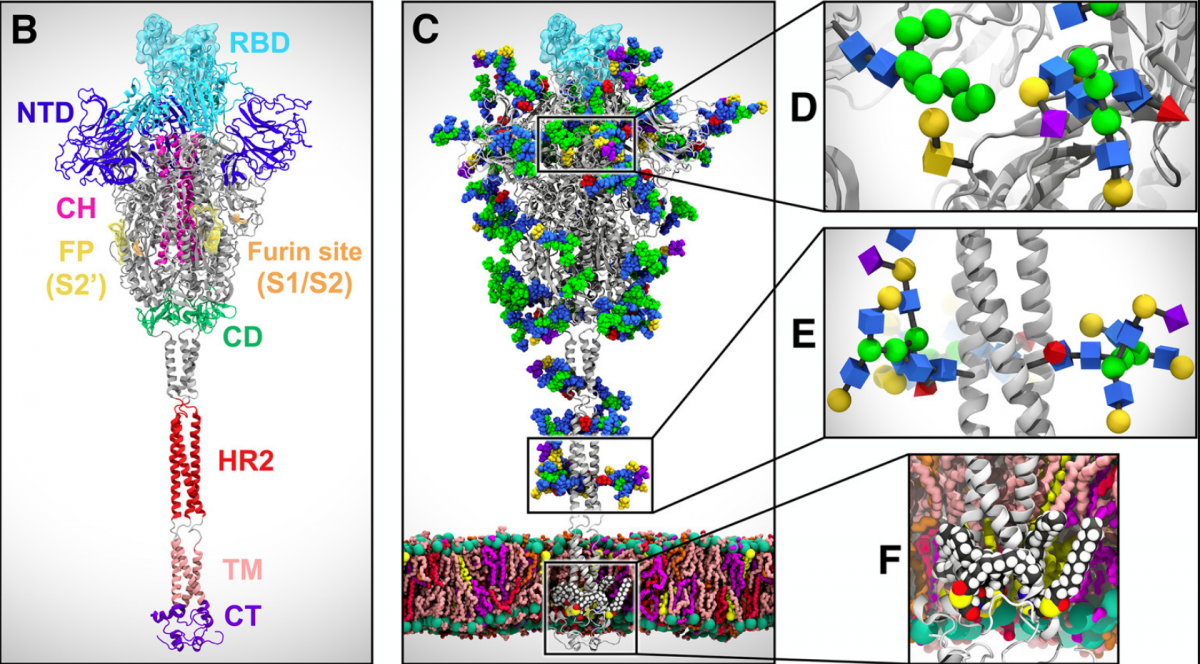
Figura 2. (B) Ensamblaje de los dominios de la proteína S en estado activo. (C) Modelo de longitud completa glicosilado y de la proteína S en estado activo incrustado en una capa bilipídica. (D,E,F)Vista ampliada de la glicosilación de la cabeza de la proteína S [1].
Fuente. Casalino, L.; Gaieb, Z.; Goldsmith, J. A.; et al. National Institutes of health. (2020)
Amaro et al. desarrollo simulaciones masivas de MD incluyendo todos los átomos del modelo glicosilado con la proteína de S del SARS-CoV-2 en un entorno donde incluye la membrana celular y el medio acuoso. El sistema de simulación abarca aproximadamente 1.7 millones de átomos que fueron simulados durante varios microsegundos incluyendo simulaciones moleculares con diferentes conformaciones de la proteína S. Esto reveló que los dos glucanos ligados a N165 y N234 no solo protegen la proteína S sino también juegan un rol estructural de modulación de la dinámica conformacional del RBD. Estos glucanos se encuentran para "bloquear y cargar" el RBD adyacente en el estado "activo" para la infección. Para probar la importancia de estos N-glicanos, se generó un mutante doble en la proteína S abierta (N165A y N243A) y se realizaron simulaciones moleculares. Como resultado, la estabilidad del RBD en el estado "activo" se ve notablemente alterada por la mutación de alanina de N165 y N234 debido a la pérdida de los respectivos glicanos unidos a N. Experimentalmente se comprobó que estas mutaciones reducen aproximadamente de 10% (N165A) y 40% (N234A) la unión de ACE2 a proteína S. Este estudio molecular además brindan una descripción completa de la dinámica de los glucanos alrededor de la proteína S. La región de la proteína S que se camufla más eficazmente con glucanos es el tallo con una cobertura total 90%. Sin embargo, la cabeza de la proteína S está menos cubierta (62%) por glucanos. Por lo tanto, al revelar estas vulnerabilidades e identificar los puntos débiles de la nanomáquina molecular del SARS-CoV-2 proporciona conocimientos sin precedentes que pueden allanar el camino para el desarrollo de nuevos fármacos contra este virus. Estos resultados refuerzan un enfoque viable para modular la plasticidad conformacional del RBD, aprovechando así la alteración de la glicosilación de la proteína S como una estrategia para el desarrollo de vacunas.
En este momento crítico, las simulaciones moleculares tienen la oportunidad de desentrañar las complejidades mecánicas del virus SARS-CoV-2 detallado a nivel atómico, ofreciendo una alternativa para estudiar de forma profunda los determinantes moleculares que juegan un papel fundamental en la virulencia del coronavirus. En la situación actual, los hallazgos de simulaciones moleculares proporcionan nuevos conocimientos sobre la proteína S del SARS-CoV-2 y su capa de glucano que pueden explotarse para el desarrollo de agentes terapéuticos contra este virus.
Bibliografía:
1. Casalino, L.; Gaieb, Z.; Goldsmith, J. A.; Hjorth, C. K.; Dommer, A. C.; Harbison, A. M.; Fogarty, C. A.; Barros, E. P.; Taylor, B. C.; McLellan, J. S.; Fadda, E.; Amaro, R. E. Beyond Shielding: The Roles of Glycans in SARS-CoV-2 Spike Protein. ACS Cent. Sci. 2020, ASAP. DOI: 10.1021/acscentsci.0c01056
2. Pablo R. Arantes, Aakash Saha, and Giulia Palermo;Molecular dynamics simulations revealed a promising immune target on the SARS-CoV-2 spike protein, proposing novel strategies for vaccine development. ACS Cent. Sci. 2020, 6, 10, 1654–1656 doi.org/10.1021/acscentsci.0c01236
3. Belouzard, S., Millet, J. K., Licitra, B. N., & Whittaker, G. R. (2012). Mechanisms of Coronavirus Cell Entry Mediated by the Viral Spike Protein. Viruses 4(6); 1011-1033. doi:10.3390/v4061011
4. Wang, Q., Wong, G., Lu, G., Yan, J., & Gao, G. F. (2016). MERS-CoV spike protein: Targets for vaccines and therapeutics. Antiviral Research 133; 165-177. doi:10.1016/j.antiviral.2016.07.015.
EN UTEC VENIMOS DESARROLLANDO LA TECNOLOGÍA
Y LA INGENIERÍA QUE NECESITA EL MUNDO DEL MAÑANA
Carreras en ingeniería y tecnología que van de la mano con la investigación y la creación de soluciones tecnológicas de vanguardia, comprometidas con las necesidades sociales y la sostenibilidad.
Decide convertirte en el profesional que el mundo necesita. Estudia en UTEC y lleva tu ingenio hacia el futuro.
Déjanos tus datos y suscríbete a nuestros boletines UTEC
-
- STUDY ABROAD
- Studying in Peru
- Living in Peru
- FAQ
- Application Form
- Contact us
-
- EXECUTIVE EDUCATION
- Executive Education
- Short Courses
- Inhouse Courses
- Calendar